![]()
Section des Sciences fondamentales de la Faculté de Biologie et de Médecine
Département de Biologie Moléculaire Végétale - Laboratoire de Phytogénétique Cellulaire
Prof. Jean-Pierre Zrÿd
Génétique III – été 2004
En collaboration avec Laurent Christinet et Andrija Finka.
Les travaux pratiques ont lieu au CP1 (205) et seront constitués de quatre expériences complémentaires :
Génétique de Brassica rapa – hybridation, incompatibilité, hérédité monoparentale.
Analyses de la ségrégation chez les hybrides du maïs Zea mays
Ségrégation des marqueurs de résistances aux antibiotiques chez des plantes transgénique de la mousse Physcomitrella patens (analyse par PCR)
Etude de l’influence des rayons ultraviolets chez le tabac Nicotiana tabacum (utilisation de gènes rapporteurs).
|
Séance 1 |
Mercredi 10 mars |
|
Séance 2 |
Mercredi 31 mars |
|
Séance 3 |
Mercredi 7 avril |
|
Séance 4 |
Mercredi 21 avril |
Les étudiants sont répartis en deux groupes : A et B. Les heures de présence aux TP sont pour le groupe A de 13h00-15h00, et pour le groupe B de 15h00-17h00.
|
Séance |
Génétique de Brassica rapa |
|
1 |
croisement par fécondation croisée de 2 génotypes différents |
|
2 |
récolte des graines |
|
3 |
semis |
|
4 |
analyse des plantes obtenues |
|
Séance |
Analyses de la ségrégation chez les hybrides du maïs Zea mays |
|
1 |
monohybridisme |
|
2 |
dihybridisme |
|
3 |
croisements test |
|
4 |
- |
|
Séance |
Ségrégation des marqueurs chez la mousse Physcomitrella patens |
|
1 |
- |
|
2 |
ségrégation des marqueurs chez Physcomitrella |
|
3 |
évaluation de la résistance aux antibiotiques et PCR de vérification |
|
4 |
analyse de la PCR |
|
Séance |
Etude de l’influence des rayons ultraviolets chez le tabac N. tabacum |
|
1 |
- |
|
2 |
- |
|
3 |
- |
|
4 |
analyse de l’expression du gène rapporteur GUS (Test GUS) |
Nous vous conseillons vivement la préparation à ces travaux pratiques disponible sur le site de l’UNIL sous : http://www.unil.ch/lpc/docs/cours1gen.htm - cliquez sur TP virtuels
Ou directement sous : http://www.unil.ch/lpc/docs/tpgen/prep/enter.htm
La variété de crucifère Brassica rapa "rapid cycling" a été sélectionnée dans le but d’obtenir des plantes avec un cycle de vie très court. Sous conditions optimales, seuls 28 jours séparent le semis de la récolte des semences. Les plantes peuvent être cultivées sur peu d’espace et les fleurs sont de dimension suffisante et donc bien adaptées à des études de fécondations (contrairement à Arabidopsis thaliana). Pour de plus amples informations on consultera le site Internet suivant :
http://www.biochem.wisc.edu/brassicaclassroomgenetics/
Le genre Brassica contient plusieurs espèces cultivées dont les choux (choux de Bruxelles, chou-fleur, brocoli et autres choux, choux-raves, etc.), la moutarde et le colza. Plusieurs de des ces espèces ont été améliorées pour obtenir des formes à cycle de vie court ; elles ont été obtenues par les méthodes de la génétique classique.
Génotypes à disposition pour vos analyses:
- Brassica rapa "rapid cycling" type sauvage (YGR/YGR ; ANL/ANL)
- ygr/ygr (yellow-green): mutation nucléaire récessive dans les voies de la biosynthèse de la chlorophylle donnant des feuilles d'un vert jaunâtre
- ygr/YGR
- anl/anl (anthocyanin-less): mutation récessive caractérisée par l’absence d'anthocyanes violets sur le bas des tiges
- anl/ANL
Des barquettes avec des plantes en fleur seront mises à votre disposition. Les plantes seront arrangées comme montrés ci-dessous.
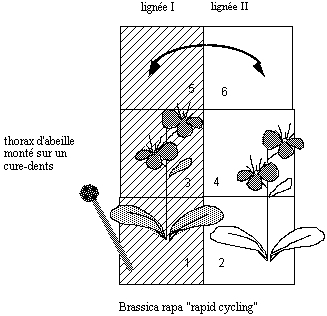
Méthode de fécondation:avec un thorax d'abeille :
Les Brassica doivent être pollinisés par des insectes. La structure du stigmate, la disposition des anthères (2 longs, 2 courts), la présence de nectarifères etc. sont parfaitement adaptés à ce but. Les abeilles sont bien adaptées à ce type de plante. Les poils sur leur corps, en particulier ceux du thorax, sont adaptés à la récolte du pollen. Aucun succédané artificiel ne remplace efficacement ce bio-matériel pour effectuer des croisements.
Croisements à effectuer: Groupe A: 1) anl/ANL x anl/anl
ou
2) anl/ANL x anl/ANL
Groupe B: 1) ygr/YGR x ygr/ygr
ou
2) ygr/YGR x ygr/YGR
Mettez une étiquette avec votre nom dans chacune de vos barquettes !
Effectuez les croisements avec les thorax d'abeilles mis à votre disposition. Pour chaque paire de plantes vous trouverez un thorax d'abeille. Commencez par la plante N°1. Prenez un thorax d'abeille, enduisez-le bien avec du pollen N°1 et fertilisez une ou plusieurs fleurs de la plante en vis-à-vis (N°2). En même temps, vous allez récolter du pollen de la plante N°2 que vous utilisez ensuite pour fertiliser la N°1. Changez d'abeille et continuez avec la paire suivante (3 & 4). Avec un autre thorax d'abeille faites le croisement entre N°5 et 6.
Les crucifères possèdent un système d'auto incompatibilité, c'est à dire qu’une plante ne peut pas être fertilisée par son propre pollen. Vous ne risquez donc pas de fausser l’expérience en utilisant plusieurs fleurs d'une même plante pour récolter du pollen, ni en passant ensuite ce thorax d'abeille sur plusieurs fleurs de la plante à fertiliser en vis-à-vis.
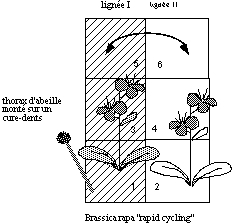
- Brassica rapa "rapid cycling" type sauvage (YGR/YGR ; ANL/ANL)
- var (variégé): mutation chloroplastique qui affecte la biosynthèse de la chlorophylle. Les plantes portent des secteurs blancs sur les feuilles
Groupe A & B: var x type sauvage
et
type sauvage x var
Pour faire les croisements, vous procéderez comme décrit précédemment.
Dans la notation génétique on note toujours le parent féminin (receveur) en premier; vous allez utiliser une fois du pollen "type sauvage" pour fertiliser une plante variégée et ensuite faire l’expérience réciproque pour vérifier comment les chloroplastes sont transmis à la descendance.
Les plantes variégées portent une mutation chloroplastique qui affecte la biosynthèse de la chlorophylle. Les plantes sont atteintes à des degrés très variables, allant de l'albinisme total jusqu'à des plantes d'apparence quasiment normale. Les secteurs blancs contiennent des cellules dont la grande majorité des chloroplastes sont dépourvus de chlorophylle, tandis que les secteurs verts contiennent des cellules dont la majorité des chloroplastes sont fonctionnels. Selon le pourcentage de chloroplastes normaux, on peut observer des secteurs d'un vert plus clair.
Chez les Brassica les chloroplastes sont hérités de la plante mère. Lors de la formation des macrospores, les chloroplastes verts et blancs sont distribués de façon aléatoire et variable. La proportion de chloroplastes albinos détermine l’intensité des la variégation. Les plantes albinos ou avec très peu de chlorophylle meurent à un stade précoce de développement. Pour effectuer les croisements on choisira des plantes avec un nombre élevé de secteurs blancs, sinon la présence de secteurs sera difficile à déceler chez la descendance.
Identifiez vos barquettes. Prenez des sachets en papier et inscrivez votre nom, la date et le croisement effectué. Stockez séparément les siliques des plantes variégées et des plantes vertes. Mettez les siliques ("gousses") dans les sachets et fermez-les. Les assistants se chargeront de mettre les graines à sécher (une semaine).
1) Ségrégation mendélienne: Dans un bac avec 48 puits, mettez 1 graine par puits et mettez une étiquette indiquant votre nom, la date et le croisement effectué.
) Hérédité uni parentale : Dans un bac avec 48 puits, mettez dans une moitié 24 graines de plantes variégées dans l'autre moitié du bac 24 graines de plantes vertes. N'oubliez pas de mettre une étiquette!
Groupe A: 1) anl/ANL x anl/anl
ou
2) anl/ANL x anl/ANL
Groupe B: 1) ygr/YGR x ygr/ygr
ou
2) ygr/YGR x ygr/YGR
Faites un schéma et prédisez la ségrégation que vous attendez.
Ensuite regardez vos plantes et introduisez vos observations dans le tableau ci-dessous. Est-ce que les résultats obtenus correspondent à la prédiction? (Effectués le test de chi-carré).
|
|
normal (vert avec anthocyanes) |
sans anthocyane |
vert jaunâtre |
vert |
|
prédiction |
a |
b |
c |
d |
|
obtenu |
a |
b |
c |
d |
Remarques ?
A quelle ségrégation vous attendez-vous?
Les résultats correspondent-ils à votre prédiction (Test du chi-carré)? Commentaires?
|
|
plantes issues de graines de la plante variégée |
plantes issues de graines du type sauvage |
||
|
variégé |
vert |
variégé |
vert |
|
|
prédiction |
||||
|
obtenu |
||||
- Quels sont les conséquences et les avantages ou inconvénients sélectifs de l'auto incompatibilité ?
- Décrire en détail les mécanismes d’auto incompatibilité dans le genre Brassica ?
- Quelles sont les stratégies possibles pour améliorer l'hétérosis chez les plantes?
- Décrivez les éléments qui font de l'abeille et des Brassica un modèle de co-évolution. Quels rôles les insectes jouent-ils dans l’évolution des plantes à fleurs?
Vous aurez un rapport à rendre sur ces travaux pratiques en génétique. Ce rapport comprendra l’ensemble des résultats expérimentaux, des calculs et remarques afférentes ; il doit aussi contenir les réponses détaillées aux questions posées. Le rapport doit être rendu au plus tard une semaine après la fin des travaux pratiques. Il vous sera remis avec les notes avant la fin du semestre.
Etudier le pollen de la F1 et les épis de la génération F2 d'un croisement, en tirer les éléments de discussion concernant la fréquence des phénotypes de divers gamètes et ses relations avec les fréquences de la génération F2 dans un cas de monohybridisme.
Le gène waxy porté par le chromosome 9 code pour l'enzyme amidon-synthase (synthèse de l’amidon). On utilisera la notation Wx (type sauvage) et wx (déficience en amidon synthase). Pour l’ensemble des caractère génétiques de ces TP on consultera l’ouvrage de Neuffer (Neuffer et al., 1997) .
![]() Déposer sur une lame de microscope une petite goutte de suspension de grains de pollen et mélanger à une goutte d’iode ioduré (Lugol – réactif de mise en évidence de l’amidon) dilué, compter les grains colorés en bleu-foncé (amidon) et ceux colorés en rouge-brun.
Déposer sur une lame de microscope une petite goutte de suspension de grains de pollen et mélanger à une goutte d’iode ioduré (Lugol – réactif de mise en évidence de l’amidon) dilué, compter les grains colorés en bleu-foncé (amidon) et ceux colorés en rouge-brun.
![]() Prendre un épi de maïs et, sans les détacher, abrasez, à l'aide de papier de verre, les téguments et la couche à aleurone de tous les caryopses jusqu'à mettre à nu l'endosperme; y déposer une goutte de Lugol. Le réactif va colorer l'amidon en bleu foncé et l'amylopectine en brun-rouge.
Prendre un épi de maïs et, sans les détacher, abrasez, à l'aide de papier de verre, les téguments et la couche à aleurone de tous les caryopses jusqu'à mettre à nu l'endosperme; y déposer une goutte de Lugol. Le réactif va colorer l'amidon en bleu foncé et l'amylopectine en brun-rouge.
Travailler par groupes de deux et compter chacun sous le microscope une préparation de pollen (au moins 100 grains chacun); ensuite compter ensemble la totalité des caryopses sur deux épis.
Tableau 1:Pollen Tableau 2: Caryopses
|
Type de pollen |
bleu foncé |
Rouge-brun |
phénotype du caryopse |
bleu foncé |
brun-rouge |
|
|
Total étudiant 1 |
Total épi 1 (N° ) |
|||||
|
Total étudiant 2 |
Total épi 2 (N° ) |
|||||
|
Total 1 & 2 |
Total 1 & 2 |
Lorsque les deux tableaux sont remplis, combiner les résultats et les classer dans les tableaux phénotypiques de la page suivante.
Lorsque les deux tableaux sont remplis, combiner les résultats et les classer dans les tableaux phénotypiques suivants:
|
phénotype du pollen |
Brun-rouge |
Bleu foncé |
Total |
|
Nb. total de grains comptés |
a1 |
a2 |
|
|
Nb. attendu selon la théorie |
e1 |
e2 |
|
phénotype des caryopses |
Brun-rouge |
Bleu foncé |
Total |
|
Nb. total de caryopses comptés |
a1 |
a2 |
|
|
Nb. attendu selon la théorie |
e1 |
e2 |
dans cet exemple la valeur du χ2 se calcule comme suit:
χ2 = (a1- e1)2 / e1 + (a2 - e2) 2 / e2
D'après vos résultats quel est le phénotype dominant? Comment êtes-vous arrivé à cette conclusion? Quelles informations pouvez-vous tirer de l'analyse des gamètes (microspores) produits par la fleur mâle du maïs?
Les épis sont une génération F2 résultant du croisement de deux lignées homozygotes pour le gène waxy, soit (Wx/Wx) et (wx/wx). Le pollen a été récolté sur la F1 de ce croisement. Pour mieux comprendre ce qui se passe, remplissez les deux tableaux suivants avec les symboles des allèles:
|
Parents |
F1 |
F2 |
||
|
Wx/Wx |
||||
|
Starchy (normal) |
___/___ ___/___ |
|||
|
X |
____/____ |
|||
|
wx/wx |
___/___ ___/___ |
|||
|
Waxy |
On voit ainsi que les gamètes de la F1 se combinent de ...... manières pour donner tous les génotypes de la F2. Ces tables doivent vous aider à remplir les tableaux 3 et 4 de la page précédente. Expliquez en détail votre démarche.
L'analyse statistique (test du χ2) vous donne des indications sur la validité de vos résultats (cf. cours de statistique). Expliquez!
Table des χ2 en fonction des degrés de liberté et de la probabilité de l'hypothèse nulle
|
d.f. |
.95 |
.90 |
.80 |
.70 |
.50 |
.30 |
.10 |
.05 |
.01 |
|
1 |
.004 |
.016 |
.064 |
.148 |
.455 |
1.07 |
2.71 |
3.84 |
6.64 |
|
2 |
.103 |
.211 |
.446 |
.713 |
1.38 |
2.41 |
4.60 |
5.99 |
9.20 |
|
3 |
.352 |
.584 |
1.00 |
1.42 |
2.37 |
3.66 |
6.25 |
7.82 |
11.3 |
d.f. = (nombre de classes-1)
Le test χ2 nous aide à prédire la probabilité p qu'une déviation donnée du résultat attendu est du uniquement à la chance, et que l'hypothèse formulée est correcte.
Exemple: avec d.f.=3 et χ2=5.2 nous nous trouvons entre .30 et .10. Donc, si l'hypothèse formulée est correcte, une déviation aussi grande se produira purement par chance dans un peu plus que 10 % des cas. En général, on met la limite à 0.05 (5%) et l'on rejette l'hypothèse si p est inférieur à cette valeur.
Etudier un ou plusieurs cas de dihybridisme en analysant les phénotypes d'une génération F2 de maïs. Se familiariser avec l'utilisation de rétro croisements (croisements test – back-crosses).
Certains caryopses sont colorés en pourpre par des pigments anthocyanidiques accumulés dans la couche à aleurone. Dans les phénotypes non colorés, c'est la couleur jaune de l'endosperme qui est visible au travers d'une couche à aleurone transparente. Le caractère pourpre ("purple") est déterminé par le gène R (dominant, pourpre) et son allèle r (incolore) portés par le chromosome 10.
Certains caryopses sont devenus ridés et plissés lors du séchage alors que d'autres ont un aspect lisse et parfaitement normal. Le caractère normal "starchy" noté Su est dominant alors que le caractère ridé et plissé su est récessif. Ces gènes sont situés sur le chromosome 4. La notation su signifie sugary/sweet et indique que le caryopse contient, peu d’amidon, mais surtout des polysaccharides solubles hautement branchés : phyto-glycogène (déficience en une enzyme de débranchage de l’amidon - liaisons α -1,6). Les caractères sont indépendants, donc chaque caryopse peut être pourpre/ridé, pourpre/lisse, jaune/ridé ou jaune/lisse.
Reporter vos résultats dans le tableau 1 et puis dans le tableau 2.
|
|
||||
|
total caryopses 1 |
||||
|
total caryopses 2 |
||||
|
total 1 & 2 |
|
|
1 |
2 |
3 |
4 |
|
|
Classes des phénotypes |
Pourpre/lisse |
Pourpre/ridé |
Jaune/lisse |
Jaune/ridé |
total 1-4 |
|
Nombre d'individus |
a1 |
a2 |
a3 |
a4 |
|
|
Nombre prédit par la théorie |
e1 |
e2 |
e3 |
e4 |
Les caryopses de la génération F2 sont le résultat d'un croisement entre deux parents P homozygotes dont les génotypes étaient (R/R Su/Su) et (r/r su/su). Quels étaient leurs phénotypes? Pour mieux comprendre, remplissez les tableaux suivants avec les symboles des allèles (R, r, Su, su) et l'indication des phénotypes correspondants.
Parents ___/___ ___/___ x ___/___ ___/___
F1 ___/___ ___/___
Croisement F1 ___/___ ___/___ x ___/___ ___/___
F2 _____ _____ _____ _____ _____ _____ _____ _____
|
____ ____ |
||||
|
____ ____ |
||||
|
____ ____ |
||||
|
____ ____ |
On voit ainsi que les gamètes de la F1 se combinent de...... manières pour donner tous les génotypes de la F2. Ces tables doivent vous aider à remplir la partie théorique du tableau 2 de la page précédente. Expliquez en détail votre démarche.
La couche à aleurone et l'endosperme sont, suite à la double fécondation typique des végétaux, des tissus triploïdes. Comment expliquez-vous alors que les résultats obtenus soient corrects ? Dans quelle mesure l'observation des caryopses est-elle représentative de ce qui se passe dans l'embryon?
L'analyse statistique (test du χ2) vous donne-t-elle des indications sur la validité de vos résultats (cf. cours de statistique) ? Expliquez !
Dans notre exemple la valeur de χ2 se calcule comme suit:
χ2 = (a1-e1)2 / e1 + (a2-e2) 2 / e2 (a3-e3)2 / e3 + (a4-e4) 2 / e4
Table des χ2 en fonction des degrés de liberté et de la probabilité de l'hypothèse nulle
|
d.f. |
.95 |
.90 |
.80 |
.70 |
.50 |
.30 |
.10 |
.05 |
.01 |
|
1 |
.004 |
.016 |
.064 |
.148 |
.455 |
1.07 |
2.71 |
3.84 |
6.64 |
|
2 |
.103 |
.211 |
.446 |
.713 |
1.38 |
2.41 |
4.60 |
5.99 |
9.20 |
|
3 |
.352 |
.584 |
1.00 |
1.42 |
2.37 |
3.66 |
6.25 |
7.82 |
11.3 |
d.f. = (nombre de classes-1)
Quel est le principe des croisements test ; quand et pourquoi sont-ils utilisés?
Etudier les différentes ségrégations obtenues et déterminer s'il s'agit d'un croisement ou d'un rétro croisement avec un parent homozygote
Certains caryopses sont colorés rouge ou pourpre par des pigments anthocyanidiques accumulés dans la couche à aleurone. Dans les phénotypes non colorés, c'est la couleur jaune ou blanche de l'endosperme qui est visible au travers d'une couche à aleurone transparente. Le trait "couche à aleurone colorée" est sous contrôle de plusieurs gènes. En plus des gènes codant pour les enzymes de la voie de biosynthèse des anthocyanes, il y a aussi des gènes régulateurs, qui peuvent activer ou inhiber l'expression des autres gènes. A vous de déterminer pour chaque cas de quel type de gène il s'agit!
Le caractère pourpre ("purple") est déterminé par le gène R (dominant, pourpre) et son allèle r (incolore) portés par le chromosome 10. Le gène C (dominant) donnant une couche à aleurone colorée, et son allèle c (incolore), sont portés par le chromosome 9. C1 (dominant) est un allèle de C et donne des caryopses incolores tandis que c1 (récessif) donne une couche à aleurone colorée.
Le gène Y (yellow) est responsable pour la coloration jaune de l'endosperme, tandis que l'allèle récessif y (white) donne un endosperme incolore.
Chaque groupe doit analyser au moins deux croisements différents.
Déterminez le nombre de caryopses présentant une couche à aleurone colorée/incolore, et parmi ceux avec une couche à aleurone incolore, comptez combien ont un endosperme jaune/blanc. Introduisez vos résultats dans le tableau 1, puis remplissez les tableaux suivants avec les phénotypes et les génotypes supposés et formulez une hypothèse pour expliquer vos résultats.
Tableau 1
|
|
aleurone colorée |
aleurone incolore |
endosperme jaune |
endosperme blanc |
|
total caryopse 1 |
Tableau 2
|
|
1 |
2 |
3 |
4 |
|
|
Classes des phénotypes |
total 1-4 |
||||
|
Nombre d'individus |
a1 |
a2 |
a3 |
a4 |
|
|
Nombre prédit par la théorie |
e1 |
e2 |
e3 |
e4 |
Les caryopses de la génération F2 sont le résultat d'un croisement entre deux parents P homozygotes dont les génotypes étaient (......../..........) et (......./.......). Quels étaient leurs phénotypes ? Pour mieux comprendre, remplissez d'abord les tableaux suivants avec les symboles des allèles et l'indication des phénotypes correspondants.
Parents ___/___ ___/___ x ___/___ ___/___
F1 ___/___ ___/___
Croisement F1 ___/___ ___/___ x ___/___ ___/___
F2 _____ _____ _____ _____ _____ _____ _____ _____
|
____ ____ |
||||
|
____ ____ |
||||
|
____ ____ |
||||
|
____ ____ |
Faites l'analyse statistique comme pour les exemples précédents. Pouvez-vous accepter ou rejeter votre hypothèse ? Expliquez votre démarche en détail.
Tableau 1
|
|
aleurone colorée |
aleurone incolore |
endosperme jaune |
endosperme blanc |
|
total caryopse 1 |
Tableau 2
|
|
1 |
2 |
3 |
4 |
|
|
Classes des phénotypes |
total 1-4 |
||||
|
Nombre d'individus |
a1 |
a2 |
a3 |
a4 |
|
|
Nombre prédit par la théorie |
e1 |
e2 |
e3 |
e4 |
Les caryopses de la génération F2 sont le résultat d'un croisement entre deux parents P homozygotes dont les génotypes étaient (......../..........) et (......./.......). Quels étaient leurs phénotypes ? Pour mieux comprendre, remplissez les tableaux suivants avec les symboles des allèles et l'indication des phénotypes correspondants.
Parents ___/___ ___/___ x ___/___ ___/___
F1 ___/___ ___/___
Croisement F1 ___/___ ___/___ x ___/___ ___/___
F2 _____ _____ _____ _____ _____ _____ _____ _____
|
____ ____ |
||||
|
____ ____ |
||||
|
____ ____ |
||||
|
____ ____ |
Faites l'analyse statistique comme pour les exemples précédents. Pouvez-vous accepter ou rejeter votre hypothèse? Expliquez votre démarche en détail.
Le gène C1 qui contrôle la coloration de la couche à aleurone a un comportement particulier. Comment l’interprétez-vous ?
Qu'est-ce qu'un gène de régulation (comme R) et pourquoi, dans ce cas-là, dit-on qu'il s'agit d'un activateur trans ?
La mousse (bryophyte) Physcomitrella patens constitue un système génétique modèle pour l'étude du développement des plantes. Comme tous les bryophytes, son cycle de vie est essentiellement haploïde, ce qui représente un avantage pour une approche génétique (mutagenèse et sélection de mutants) de l'étude de certains métabolismes(Ashton and Cove, 1977). Après fertilisation de l’oocyte par des spermatozoïdes (en milieu aqueux), le zygote se développe en petit sporophyte diploïde. La méiose se produit dans la capsule menant à la formation des spores (approximativement 5.000 spores par capsule).
Dans notre laboratoire nous avons démontré que l’intégration d’ADN étranger par recombinaison homologue avec des séquences définies du génome de P. patens (ciblage de gène - gene targeting) est au moins 1000 fois plus efficace que chez les plantes supérieures, ce qui permets de réaliser directement et pour la première fois sur une plante des expériences de knock-out et de mutagenèse ponctuelle (Schaefer and Zryd, 2001; Schaefer, 2002). P. patens est auto-fertile et des croisements-test ont été facilités par l'utilisation des mutants auxotrophiques mâle-stériles.
Le but des expériences proposées dans ces TP est de réaliser comment fonctionne la ségrégation génétique dans un organisme haploïde. La première transformation réussie a été réalisée par transfert d'ADN dans les protoplastes avec les plasmides portant les marqueurs pour la résistance sur antibiotiques hygromycine et kanamycine que permet de sélectionner les souches transformées (Schaefer, 1994). La souche mâle-stérile est auxotrophe pour l’acide nicotinique et déficiente en chlorophylle (couleur jaune) et elle peut être identifiée facilement dans les expériences des croisements. Les souches de mousses résistantes à l’ hygromycine et à la kanamycine (transgénique double résistants) sont croisées avec la souche nicB5ylo6 et les spores collectées sont mises à germer les plantes analysées après environ trois semaines de culture sur un milieu non sélectif.
Remplissez le tableau suivant :
|
génotypes des gamétophytes |
génotype du sporophyte sur plante nicB5ylo6 (jaune) |
Génotypes des spores |
|
HygR , KanR Normal (vert) |
___ ,____,___________, ___ ,____,___________, ___ ,____,___________, ___ ,____,___________, ___ ,____,___________, ___ ,____,___________, ___ ,____,___________, ___ ,____,___________, |
|
|
X |
___ /___ , ___ /___ , ___ /___ / |
|
|
HygS, KanS nicB5ylo6 (jaune)* |
* NB : mâle stérile
Votre tâche est de repérer les colonies (environ 100) dérivant d’une spore unique, de les partager en deux fragments et de mettre ceux-ci en culture ; l’un sur un milieu avec hygromycine et l’autre sur un milieu avec kanamycine.
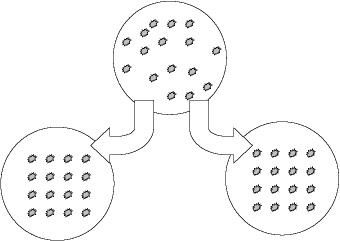
Hyg 30 mg/l ---------------------------------- Kan 50mg/l
Déterminez après sept jours les fréquences de clones résistants sur hygromycine, kanamycine, ou sur les deux, ainsi que les clones sensibles aux antibiotiques. Après 20 jours déterminer la fréquence des clones jaunes parmi les résistants.
|
|
plantes vertes |
plantes jaunes |
||||||
|
HygR KanR |
HygS KanR |
HygR KanS |
HygS KanS |
HygR KanR |
HygS KanR |
HygR KanS |
HygS KanS |
|
|
obtenu |
||||||||
|
prédiction |
||||||||
Faites l'analyse statistique comme pour les exemples précédents. Pouvez-vous accepter ou rejeter votre hypothèse?
Réaction de polymérase en chaîne (PCR) est la technique la plus puissante pour faire détection l’ADN en faible quantité (dans l’ordre du picogramme ; 10-12g). Ainsi on peut détecter la présence du gène de marquer dans l’ADN génomique (déjà isolé par l’assistant) utilisant des amorces spécifiques. Dans le schéma suivant vous pouvez voir le placement des gènes responsable pour la résistance sur antibiotique avec ses amorces correspondantes
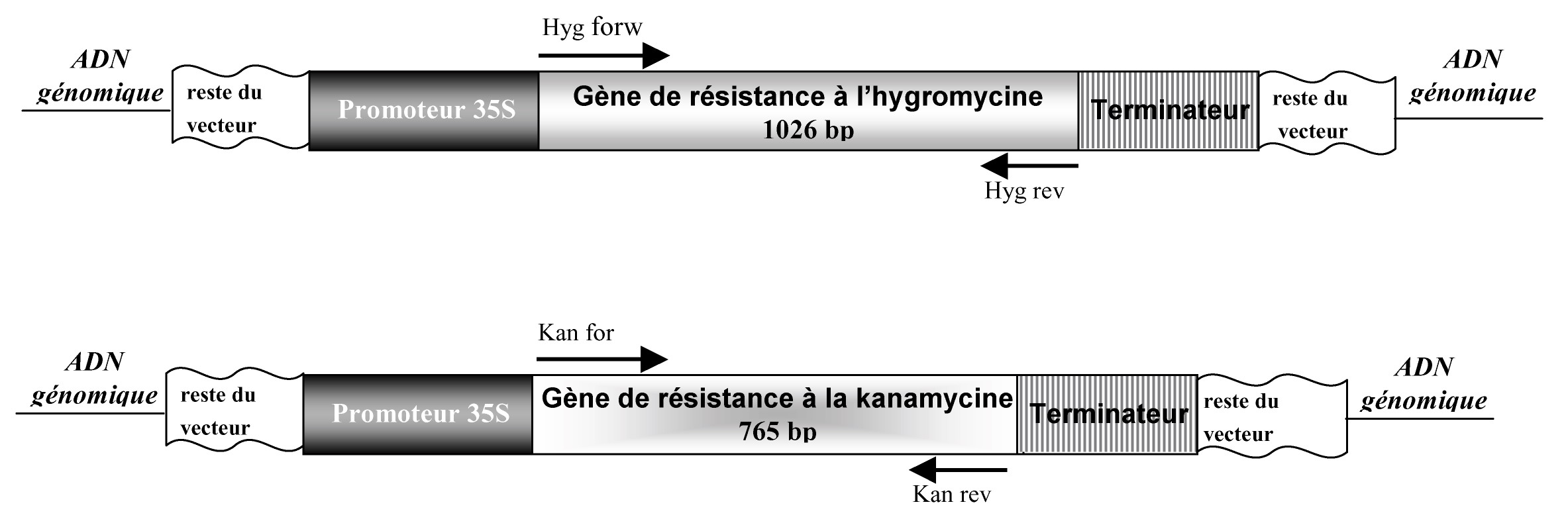
Nous avons préparer la réaction PCR de la façon suivante :
Mélangez :
5 μl de tampon PCR
2.5 μl de MgCl2 50 mM
1 μl d’amorce marqué for
1 μl d’amorce marqué rev
1 μl d’amorce 10 mM dNTP
x μl d’ADN correspondant ~200ng au total
0.5 μl de polymérase Taq (INVITROGEN)
ajoutez de l’eau jusqu’au volume final de 50 μl
Les cycles thermiques de la PCR doivent être programmés comme suit :
4’ @ 94°C - dénaturation initiale d’ADN
1’ @ 94°C - dénaturation d’ADN par cycle
40 cycles
1’ @ 50°C - « annealing » des amorces
2’ @ 72°C- polymérisation
Les résultats de cette expérience (photo du gel) seront mis à votre disposition sur le site Internet du laboratoire http://www.unil.ch/lpc/docs/cours1gen.htm#TP
Est-ce qu’est votre analyse moléculaire correspond à votre analyse génétique expliquer en détail les arguments justifiants votre réponse?
Que pouvez-vous conclure de la ségrégation des différents marqueurs chez mousse ?
Décrivez en détail le cycle de vie chez un bryophyte, en particulier décrivez la reproduction, l’alternance des générations et ses conséquence sur l’analyse génétique !
De nombreux facteurs externes tels que les rayonnements ultraviolets, les radiations ionisantes ou certains composés chimiques mutagènes provoquent de graves altérations au niveau de l’ADN des organismes vivants. Ces altérations correspondent souvent à des cassures au niveau des brins de l’ADN, engendrant une augmentation des possibilités de réarrangement au niveau de l’ADN, notamment par recombinaison homologue intrachromosomale.
Nous allons étudier l’influence des rayonnements ultraviolets (UV) sur des plants de tabac entier dans des conditions expérimentales proches de celle rencontrées dans des conditions naturelles. A cette fin, nous utilisons une variété de tabac transformé contenant le gène rapporteur de la β-glucuronidase (GUS) interrompu en son milieu par un gène de résistance à l’hygromycine (Swoboda et al., 1994). Le gène GUS est donc inactif. L’exposition aux UV peut, sous certaines conditions, engendrer une cassure entre les deux fragments du gène GUS dont la fonction pourra alors être restaurée par recombinaison homologue (Ries et al., 2000).
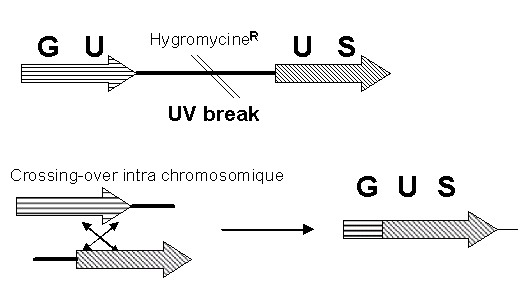
Les cellules dans lesquelles la recombinaison aura eu lieu possèderont à nouveau un gène GUS fonctionnel ; elles exprimeront donc le produit de ce gène l’enzyme β-glucuronidase. Sa présence au niveau des tissus végétaux peut être mise en évidence indirectement en présence d’un substrat spécifique de l’enzyme le 5-bromo-4-chloro-3-indolyl glucuronide. Le produit de la réaction d’hydrolyse est bleu ; il est ainsi possible de quantifier les fréquences de recombinaison, chaque tache correspondant à un événement de recombinaison indépendant.
Ces plantes ont été utilisées comme bio indicateur de pollution par des radiations ionisante, suite à des accidents de centrales nucléaires par exemple (Kovalchuk et al., 1998). Grâce au système de gène rapporteur GUS étudié ici, il est donc possible de quantifier l’impact d’une pollution ionisante sur un milieu. Ceci peut être très pratique, afin d’évaluer la progression d’une décontamination d’un milieu en vue de sa recolonisation.
Matériel végétal :
Plantules de tabac (Nicotiana tabacum) transformées N1C4 #9 possédant le gène de la β-glucuronidase GUS modifié et un gène de résistance à l’hygromycine.
Tampon de coloration histochimique GUS :
Na2HPO4/ NaH2PO4 (NaPi) 0.2 M
K3[Fe(CN)6] 0.5 M
K4[Fe(CN)6]3H2O 0.5 M
Na2EDTA 0.01 M
5-bromo-4-chloro-3-indolyl glucuronide 0.1 mg/ml
pH 8.0
Des plantules de tabac N1C4 #9, possédant le gène de la β-glucuronidase GUS modifié, sont mises à germer sur un milieu additionné d’hygromycine (50 mg/l) durant 10 jours. Elles sont ensuite transférées sur le même milieu sans antibiotique et après quelques jours subissent différentes expositions aux UV.
pas d’exposition aux UV
exposition courte aux UV (par exemple 10 s)
exposition prolongée aux UV (par exemple 30 s)
Vous recevrez ces plantules âgées de 6 semaines. Vous devez les prélever et les immerger 2 heures à 37°C dans le tampon GUS contenant le substrat de la β-glucuronidase afin de révéler histochimiquement la présence de l’enzyme. Une décoloration de la chlorophylle en présence d’éthanol est effectuée durant une nuit pour permettre de mieux distinguer les zones bleues d’activité enzymatique. Un comptage des cellules colorées en bleu et du nombre d’individu vivant sera fait pour chaque population le lendemain (une population par traitement).
Vous présenterez un tableau détaillé du nombre de plantes viables, du nombre de tache par plante, etc.… en fonction de la durée d’exposition aux UV.
Y-a t’il un effet d’une exposition au rayonnement UV ?
Quels sont les mécanismes de réparations de l’ADN mis en œuvre par une plante à la suite d’une exposition à des UV ?
Quel est l’effet de la lumière visible sur un organisme exposé aux UV ? Que se passe-t-il si cet organisme est maintenu pendant quelques heures à l’obscurité ?
Quel est l’intérêt de la recombinaison homologue par rapport à la recombinaison hétérologue – a) : pour la plante, b) : dans la perspective d’une utilisation biotechnologique ?
Ashton NW, Cove DJ (1977) Isolation and Preliminary Characterization of Auxotrophic and Analog Resistant Mutants of Moss, Physcomitrella-Patens. Molecular & General Genetics 154: 87-95
Kovalchuk I, Kovalchuk O, Arkhipov A, Hohn B (1998) Transgenic plants are sensitive bioindicators of nuclear pollution caused by the Chernobyl accident. Nature Biotechnology 16: 1054-1059
Neuffer MG, Coe EH, Wessler SR (1997) Mutants of Maize. Cold Spring Harbor Laboratory Press
Ries G, Heller W, Puchta H, Sandermann H, Seidlitz HK, Hohn B (2000) Elevated UV-B radiation reduces genome stability in plants. Nature 406: 98-101
Schaefer DG (1994) Molecular genetic approaches to the biology of the moss Physcomitrella patens. PhD Thesis, University of Lausanne, Switzerland "www.unil.ch/lpc/docs/DSThesis.htm"
Schaefer DG (2002) A newmoss genetics: Targeted mutagenesis in Physcomitrella patens. Annual Review of Plant Biology 53: 477-501
Schaefer DG, Zryd JP (2001) The moss Physcomitrella patens, now and then. Plant Physiology 127: 1430-1438
Swoboda P, Gal S, Hohn B, Puchta H (1994) Intrachromosomal Homologous Recombination in Whole Plants. EMBO Journal 13: 484-489